El pasado martes tuve el placer de impartir el curso “Fundamentos metodológicos de revisiones sistemáticas”, organizado por BiblioSalud, la red de bibliotecarios y profesionales de la información en ciencias de la salud de España. Esta sesión formativa, dirigida especialmente a bibliotecarios médicos, se centró en los aspectos clave que debemos conocer para comprender y acompañar con rigor el desarrollo de una revisión sistemática.
En esta entrada del blog quiero comenzar a compartir una selección de las herramientas y recomendaciones prácticas que utilizo habitualmente en mi trabajo diario con documentos de síntesis de evidencia.
El objetivo es ofrecer una guía no exhaustiva, hay muchas y variadas (ver figura) pero con recomendaciones y herramientas útiles y basada en mi experiencia, que sirva tanto a quienes empiezan como a quienes ya participan en revisiones sistemáticas desde las bibliotecas.
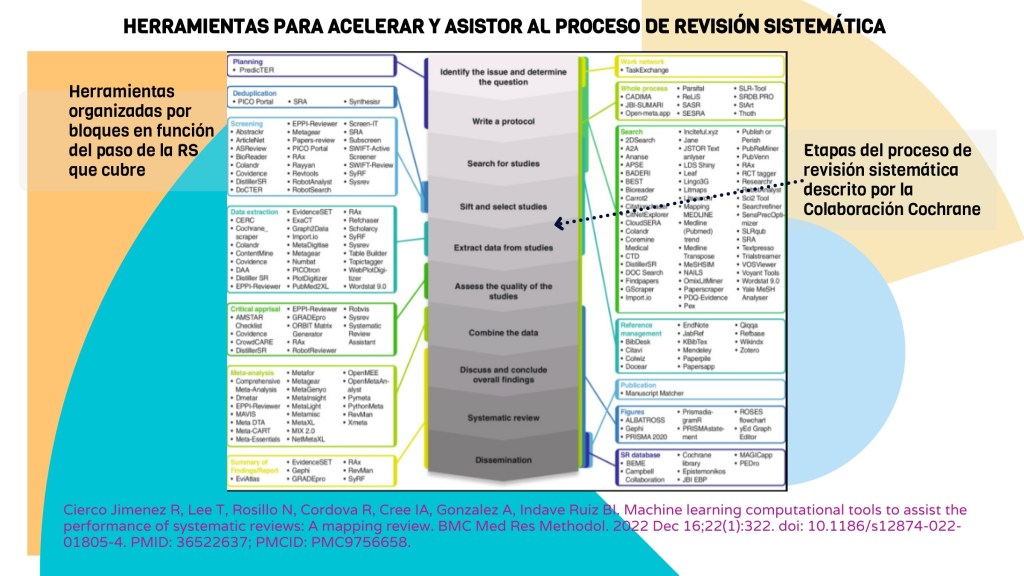
Voy a dividir las herramientas en varios apartados según su uso:
- Recolección de términos controlados y libres.
- Proceso de búsqueda y cribado.
- Chatbots e inteligencia artificial para apoyar la confección de estrategias booleanas.
- Herramientas para la búsqueda complementaria.
- Otras herramientas recomendadas para las revisiones sistemáticas.
En esta entrada veremos las primeras de estas herramientas y recomendaciones para la recolección de términos.
Recolección de términos
Uno de los pasos más críticos en cualquier revisión sistemática es la correcta identificación de términos controlados y palabras clave. Una estrategia de búsqueda bien construida comienza con una recolección exhaustiva de términos y para ello contamos con una serie de recursos imprescindibles:
1.1. Entry Terms de MeSH Database.
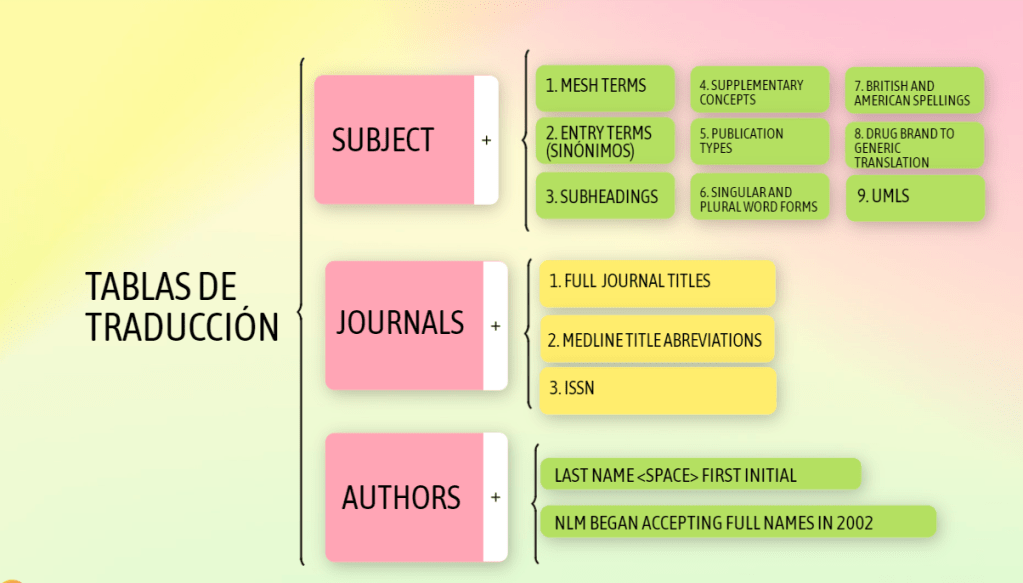
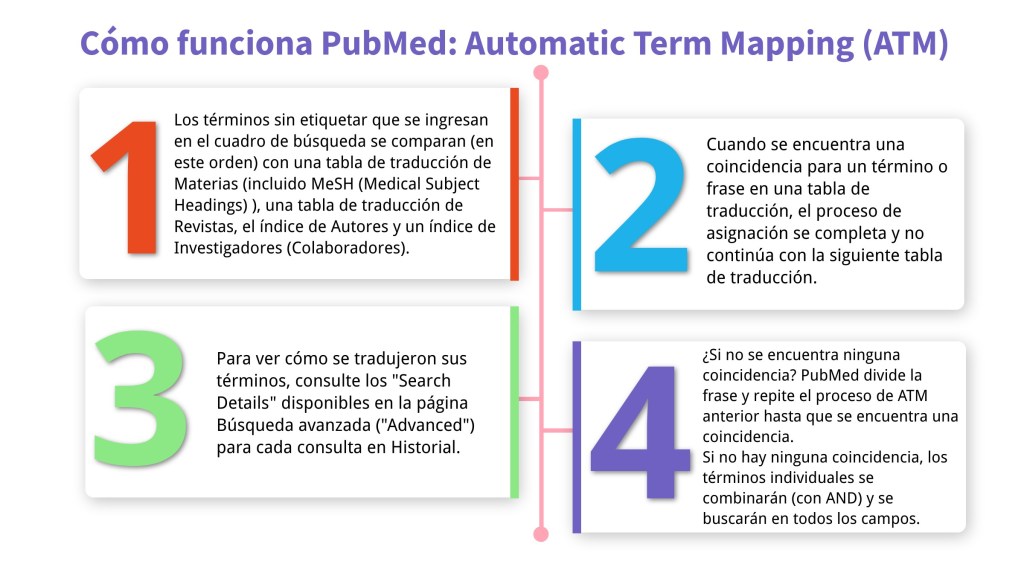
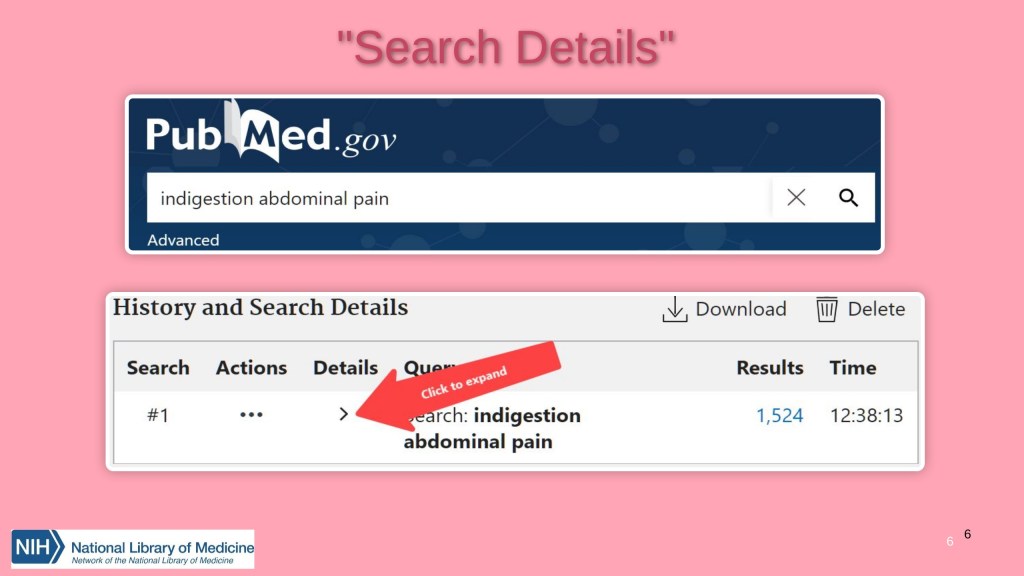
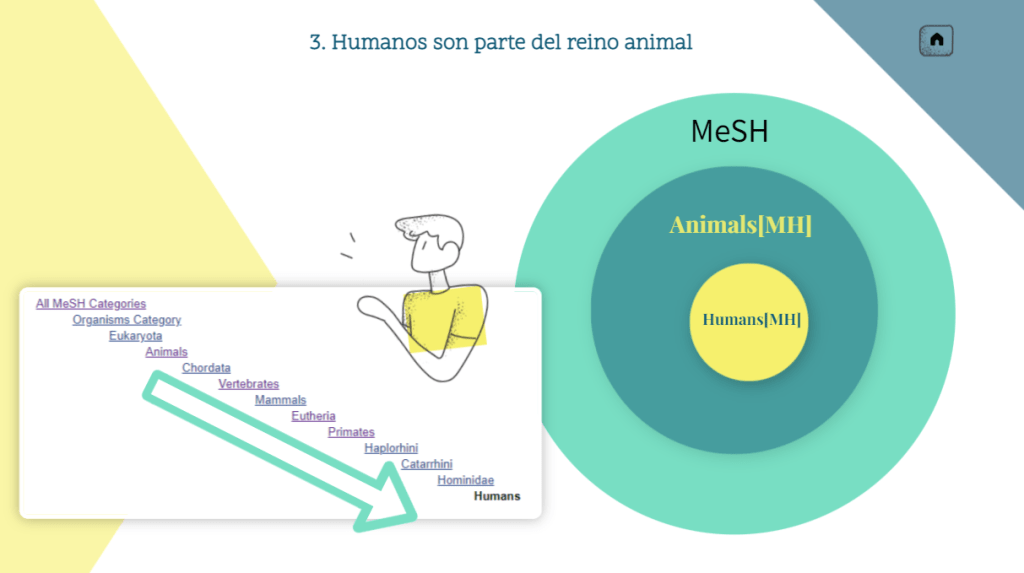
La herramienta clásica para explorar la jerarquía de los Medical Subject Headings (MeSH). Nos permite ver la definición de cada término, sus sinónimos, su posición dentro del árbol jerárquico, subencabezamientos permitidos y términos relacionados o «Entry Terms». Fundamental para identificar los términos controlados exactos que se deben utilizar en PubMed y para entender el enfoque conceptual con el que el sistema indexa los documentos.
1.2. Sinónimos de Emtree de EMBASE
El Emtree es el tesauro específico de la base de datos Embase. Su lógica es similar al MeSH pero tiene su propia estructura y contiene más términos, especialmente útiles para temas en farmacología o dispositivos médicos. Conviene hacer búsquedas paralelas en MeSH y Emtree para comparar coberturas y encontrar sinónimos o variantes terminológicas que pueden enriquecer la estrategia.
1.3. Yale MESH Analyzer https://mesh.med.yale.edu/
Esta herramienta permite comparar los términos MeSH asignados, los términos empleados en el título y resumen así como las palabras clave de los autores a un conjunto de artículos. Es especialmente útil para detectar patrones o términos recurrentes en la literatura más relevante sobre tu tema. Solo necesitas extraer los PMIDs de los artículos clave y el analizador los agrupa mostrando visualmente los MeSH comunes. Ideal para verificar si estás pasando por alto términos relevantes o para ajustar tu estrategia inicial.
1.4. Word Freq de SR Accelerator (ahora TERA) https://tera-tools.com/
Esta herramienta permite analizar la frecuencia de palabras en títulos y resúmenes de los resultados de una búsqueda bibliográfica. Solo necesitas copiar y pegar el listado de referencias (por ejemplo, desde PubMed) y Word Freq generará un listado de términos ordenados por frecuencia. Es especialmente útil para:
- Identificar términos clave y sinónimos que puedes haber pasado por alto.
- Detectar conceptos emergentes o patrones de lenguaje en la literatura.
- Afinar tu estrategia de búsqueda ajustando los términos libres.
Una herramienta sencilla pero muy eficaz para enriquecer la fase exploratoria de cualquier revisión sistemática.
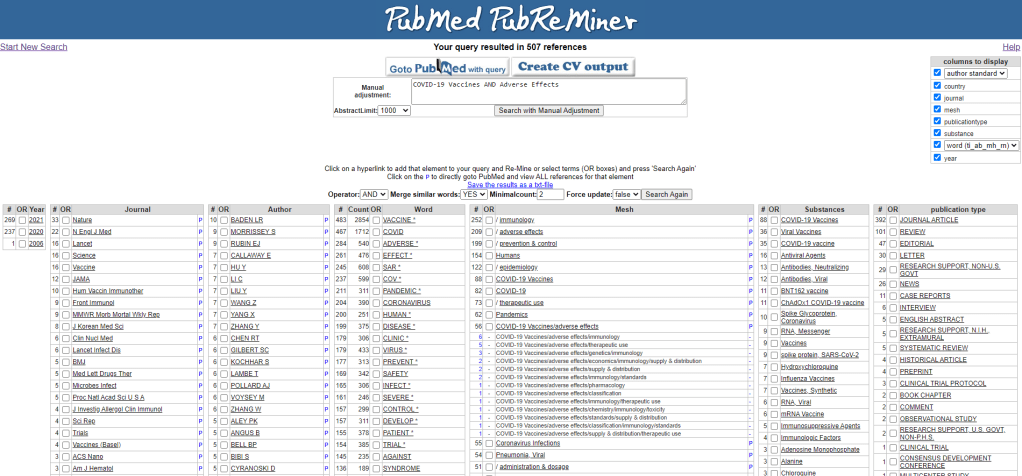
1.5. PubReminer https://hgserver2.amc.nl/cgi-bin/miner/miner2.cgi
Es una potente herramienta para realizar minería de datos sobre resultados de PubMed. Introduciendo una lista de PMIDs o haciendo una búsqueda directa, puedes ver las frecuencias de palabras en títulos, resúmenes y términos MeSH. También puedes analizar la aparición de autores, revistas, años de publicación… Es perfecta para afinar términos libres y obtener una imagen más clara del contexto bibliográfico.
1.6. MeSH on Demand https://meshb.nlm.nih.gov/MeSHonDemand
Desarrollado por la National Library of Medicine, este recurso es especialmente útil cuando partimos de un texto breve (como el resumen de un protocolo o el título de un artículo). Solo tienes que copiar el contenido y MeSH on Demand te devuelve sugerencias de términos MeSH relevantes. Es una excelente forma de inspirarte cuando aún estás perfilando la estrategia inicial.
1.7. Asistente de inteligencia artificial MeshMaster
MeSHMaster es un chatbot especializado que utiliza inteligencia artificial para ayudarte a construir estrategias de búsqueda booleanas, combinando términos MeSH y palabras clave libres. A partir de una descripción breve del tema o una pregunta clínica, el asistente sugiere:
- Términos MeSH relevantes y sinónimos.
- Combinaciones booleanas iniciales (AND, OR, NOT).
- Variantes terminológicas en inglés que pueden enriquecer la sensibilidad de la búsqueda.
Es una herramienta especialmente útil en las primeras fases de diseño de la estrategia, cuando se busca inspiración o una validación rápida de conceptos clave. Aporta rapidez sin perder la lógica estructural que requiere una buena estrategia de búsqueda.
Te invito a seguir mis próximas entradas para descubrir cómo integrar estas herramientas en tu revisión sistemática.